TD 4 - Mean-Shift clustering et segmentation d'images
(application des Kd-trees)
Introduction:
Dans ce TD nous allons implanter et tester un certain nombre de
méthodes faisant intervenir des algorithmes de recherche
géométrique (en dimension générique k). Pour faciliter votre travail, un certain
nombres de primitives pour la manipulation de points en dimension
kD (k arbitraire), ainsi que des
classes pour la manipulation d'images en Java, seront fournies au
cours de ce TD.
Fichiers et documentation pour le TD d'aujourd'hui:
- La documentation de la bibliothèque Jcg est
consultable ici
- les squelettes des classes
pour ce TD
- données pour tester (fichier InputDataImages.zip):
nuages de points en 2D et 3D, et images de test.
- voici la documentation pour
ce TD.
- la nouvelle version de la librairie Jcg (à sauvegarder): Jcg.jar
Mean-shift clustering: une petite introduction
Dans ce TD nous considérons un algorithme de clustering connu
sous le nom de Mean-Shift: en particulier, nous verrons une
application au problème de la segmentation d'images.
Segmentation d'images
La classe ImageManipulation
met à disposition un certain nombre de méthodes pour manipuler des
images en différentes formats, comme:
- l'ouverture d'une image à partir d'un fichier (au format jpg,
png, bmp, ...),
- des méthodes pour la manipulation des couleurs des pixels de
l'image
- ...
Segmentation d'images et clustering
Segmentation
L'idée de base est très simple: on part d'une image de dimension
NxM pixels, et on envoie chaque pixel sur un point dans un espace
de couleurs de dimension 3 (ici espace Luv). On obtient ainsi un
nuage de N.M points dans R^3. On applique un algorithme de
clustering (Mean-Shift aujourd'hui) sur ce nuage afin de
determiner une classification des points (et donc des pixels de
l'image). Une fois les clusters calculés, on associe à tous les
points (pixels) d'un meme cluster la meme couleur: cette phase est
déjà codée et mise en place par la classe ImageSegmentation.
Clustering
(Idée générale: plus de détails dans la suite) On traite
l'ensemble des points comme provenant d'une fonction de densité de
probabilité (qu'on ne connait pas explicitement, bien sur): les
régions de l'espace où la densité est forte correspondent aux maximaux locaux (modes)
de la distribution sous-jacente. L'algorithme de clusterting
appelé Mean-Shift
consiste à effectuer des estimations locales du gradient de la
densité aux points de données, puis à bouger ces points le long du
gradient estimé de manière itérative, jusqu'à ce qu'il y ait
convergence: les points
stationnaires de ce procedé correspondent aux maximaux
locaux de la distribution. Et finalement, les points qu'on associe
à un meme point stationnaire seront classifiés comme faisant
partie du meme cluster.
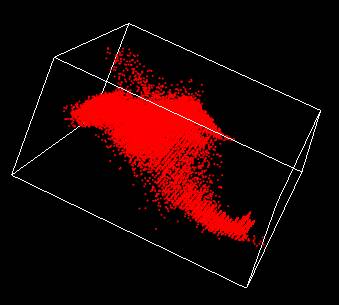
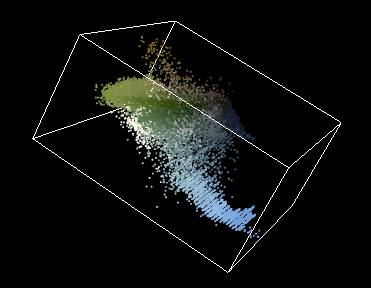
Voici un exemple:

|
|
|

|
Image originale
(260x195 pixels)
|
Points dans l'espace
Luv (3 dimensions)
|
Après calcul des
clusters avec Mean-Shift (le paramètre de
bandwidth est fixé a 2.2, voir la section 2) |
Après segmentation
|
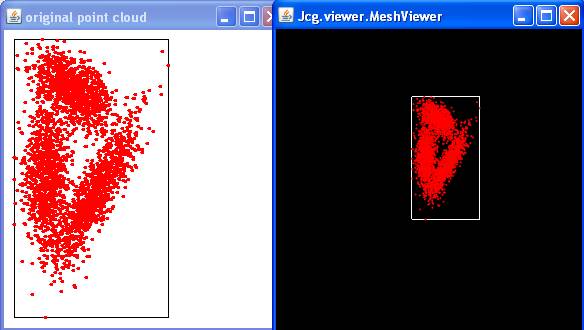
0. Pour commencer, quelques tests
Ouverture et affichage d'une image
On va commencer par effectuer des tests à l'aide de la classe TestIO. Pour
executer le programme il faut lui passer en argument le nom d'un
fichier image (.jpg, .bmp, .png) ou bien un fichier au contenant des
nuages de points (.dat).
Voici les resultats qu'on peut obtenir.


|
|
java TestIO data/EP.jpg
|
java TestIO data/2D_dataset1.dat
|
Comment résoudre cet TD: en suivant des consignes... ou en
partant de rien (selon vos gouts)
éléments dont vous disposez au cours de ce TD (un petit résumé)
Structures de données
fournies
|
RangeSearch, FastRangeSearch,
SlowRangeSearch |
Implémentations éfficaces et
naives de Range Queries
|
|
|
PointCloud
|
Nuage de points:
implementation à base de listes chainées
|
|
|
CoordinateComparator
|
Un comparateur permettant de
comparer les valeurs de la i-ième coordonnée de deux points
(utile pour trier un tableau (exo 1.1b), avec la
méthode: Arrays.sort()
)
|
|
Manipulation d'images et
données
|
Draw
|
Méthodes pour la
visualization de nuages de points en 2D et 3D
|
|
|
ImageManipulation (pour
tester exo 0)
|
Ouverture de fichiers images,
calcul d'un nuage de points à partir d'une image (et
viceversa)
|
|
|
Clustering
|
Permet de manipuler des
nuages de points et des clusters (ouverture de fichiers,
...)
|
|
|
|
|
|
Fichiers
à
compléter
|
KdTree (exo 1)
|
Implementation d'un Kd-Tree
et du calcul des plus proches voisins
|
|
|
MeanShiftClustering (exo 2) |
Implementation de
l'algorithme Mean-Shift |
|
|
LinearTimeMedian (facultatif)
|
implémentation de
l'algorithme linéaire pour le calcule de la médiane
|
|
|
|
|
|
Fichiers
de test
|
TestIO (test exo 0) |
tests préliminaires sur les
données en entrées
|
|
|
TestMedian (exo 1)
|
Test pour le calcul de la médiane
|
|
|
TestKdTree (exo 1)
|
Test sur la construction du Kd-Tree
|
|
|
TestRangeSearch |
Test la recherche des points proche avec
Kd-tree (range search)
|
|
|
TestMeanShift (test exo 2)
|
Test l' algorithme Mean Shift
|
|
|
TestImageSegmentation (test exo2) |
Permet d'appliquer le clustering à la
segmentation d'images |
|
Des squelettes à compléter (avec tests)
|
En partant de rien (ou presque)
|
|
Comme autrefois, on vous suggère de
compléter les squelettes des fonctions (code déjà fourni).
Dans ce cas, pour chaque exercice il vous reste à
compléter le squelette d'une fonction, ainsi qu'à tester
votre code avec des méthodes déjà fournies.
Et plus précisément, on vous demande de résoudre les
questions dans cet ordre:
Exo 1: fichier KdTree.java
- 1.1: calcul du point median d'un nuage de point (class
LinearTimeMedian() )
- 1.2: construction récursive d'un Kd-Tree, pour un
nuage de points donné (constructor KdTree(PointCloud N, int pDim, int cutDim)
)
- 1.3: calcul des voisins d'un point
donné: les points se trouvant à distance au
plus R d'un point donné (fonction OrthogonalRangeSearch(Point_D
q,
double sqRad) )
Exo 2: fichier MeanShiftClustering.java
- 2.1: détection d'un cluster
- 2.2: fusion d'un cluster donné avec les autres déjà
existants (se trouvant proche de ce cluster)
- 2.3: détection de tous les clusters
Exo 3: fichier TestImageSegmentation.java
- 3.1: application de Mean-Shift à la segmentation
d'images (pas de code à écrire):
effectuer des tests
- 3.2: implementation d'une petite optimization pour le
calcul du clustering (fichier MeanShiftClustering.java)
|
Dans ce cas on vous laisse plus de
liberté dans la conception des structures de données et
algorithmes: c'est à vous de choisir et mettre en place
les structures que vous trouverez plus convenables pour
une resolution éfficace des problèmes posés.
Bien sur, dans ce cas les méthodes fournies pour effectuer
les tests ne sont pas censées fonctionner correctement: il
vous faudra peut-etre les adapter.
On vous demande d'écrire deux classes comme suit:
Exo 1: Fournir la classe KdTree des méthodes
- public static KdTree constructDataStructure(PointCloud
N,
int pDim)
qui construit et renvoie un KdTree, correspondant à un nuage
de point donné, en dimension pDim.
- public PointCloud NearestNeighbor (Point_D q,
double sqRad)
qui calcule et renvoie la liste des points qui sont à
distance au plus squRad du point q (point de requete).
Exo 2: fournir la classe MeanShiftClustering de
- des données et paramètres
suivants
- N: nuage de points (input)
- sqCvgRad: rayon de
convergence (au carré)
- sqAvgRad: rayon de la
Fenetre (au carré)
- sqInflRad: rayon
d'influence (au carré)
- sqMergeRad: distance de
fusion (au carré)
- public MeanShiftClustering
(PointCloud n, double bandWidth)
le constructeur qui initialise la classe MeanShiftClustering
(déjà fourni dans le squelette).
- public Point_D[] detectClusters
()
qui met en place l'algorithme de clustering appelé
Mean-Shift:
Exo 3: fichier TestImageSegmentation.java
- 3.1: application de Mean-Shift à la segmentation
d'images (pas de code à écrire):
effectuer des tests
- 3.2: implementation d'une petite optimization pour le
calcul du clustering (fichier MeanShiftClustering.java)
|
|
|
1. Creation et manipulation des Kd-trees
Dans cet exercice on vous demande de compléter
la classe KdTree: comme vu en cours aujourd'hui, les
kD-trees permettent de répondre efficacement à des requetes de
proximité géométrique, telles que le calcul des plus proches
voisins (Nearest Neighbor Search) ou les "range search
queries".
L'idée à la base de cet approche est très simple: on construit une
décomposition récursive de l'éspace comme dans l'exemple
ci-dessous.
|
Construction:
La construction de l'arbre (binaire) se réalise en
effectuant un partitionnement récursif d'un nuage de points
N à l'aide
d'hyperplans séparateurs.
Les kD-trees sont des arbres binaires définis comme suit:
- tout noeud est associé à un hyper-rectangle et
contient les informations suivantes:
- le nuage de points
contenu dans l'hyper-rectangle (défini par la classe PointCloud)
- les informations concernant le plan de coupe: la direction, ainsi
que sa position
(par construction le plan séparateur passe par la
médiane)
- les feuilles contiennent des points isolés (des PointCloud
de taille 1)
- la racine est le nuage de points initial N.
- pour tout noeud de l'arbre, ses descendants sont les
deux sous-graphes obtenus avec découpage à l'aide d'un
plan séparateur (qui est, par construction, parallèle
aux axes des coordonnées).
Recherche
des
plus proches voisins d'un points donné (à distance au plus
R):
Le calcul des voisins d'un point donné
q (plus proches d'une
distance donnée), s'effectue récursivement à l'aide d'une
visite de l'arbre (en partant de la racine).
A chaque étape il faut tester si le disque centré en q, et de rayon R, intersèctent ou pas le
plan séparateur:
-
si le disque intersècte le plan
séparateur, alors il faut continuer la recherche
récursivement dans les deux sous-arbres (gauche et
droit): le résultat de la requete sera alors l'union des
deux requetes récursives dans les deux sous-arbres
-
s'il n'y a pas d'intersection avec
le disque séparateur, alors cela signifie que la
recherche ne sera effectué que dans un seul sous-arbre
(celui dont le rectangle correspondant contient le
point q):
dans ce cas, il faut determiner si le disque se trouve
à "droite" ou à "gauche" du plan séparateur.
|
|
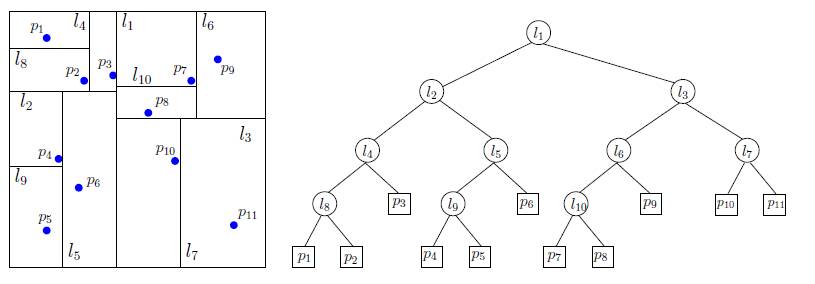
(exemple de 2d-Tree)
|
1.1 Calculer l'hyperplan séparateur
Le choix de l'hyperplan separateur (parallèle
aux axes) peut se faire de plusieurs manières différentes.
La direction du
plan séparateur sera dénoté par une valeur cutDim,
dans la suite: en dimension d, cutDim vaut 0 ... d-1.
Dans ce TD, on vous propose 3 méthodes: de préférence, on vous demande
d'utilise celle basée sur le calcul de la mediane avec un
algorithme de complexité linéaire.
Les images ci-dessous illustrent les trois méthodes possibles.
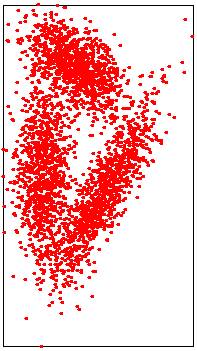
|
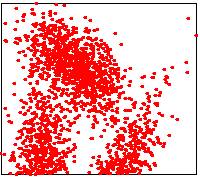

|
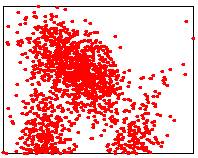
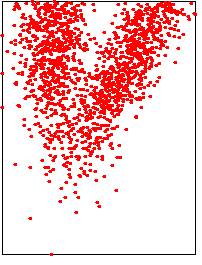
|
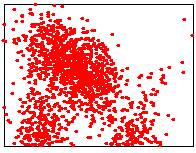
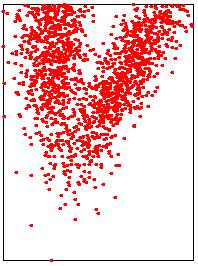
|
|
Input point cloud
|
Mediane en temps linéaire
|
Mediane de points triés (nlog
n)
|
Avec barycentre (temps
linéaire)
|
Médiane linéaire: points aléatoires
|
|
Lower points: 1214/3000 |
Lower points: 1500/3000 |
Lower points: 1596/3000
|
Lower points: 1489/3000 |
1.1a Calculer la médiane en temps linéaire (facultatif)
|
voici quelque résultats
|
|
Pour le calcul de la médiane on vous
suggère de mettre en place l'algorithme de complexité
linéaire vu en cours aujourd'hui seulement s'il vous
reste de temps à la fin du TD.
Vous avez à compléter les deux fonctions suivantes:
- private static Point_D findMedianArray (Point_D[]
buffer, int max, int cutDim)
qui renvoie la médiane d'un tableau de taille max, pour des
tableaux de petite taille (max sera au plus 5, dans la
pratique). Le paramètre cutDim indique la direction de la
coupe.
- private static Point_D findMedianLinear (PointCloud
N, int cutDim)
qui renvoie la médiane des cutDim-èmes coordonnées du
nuage de points N. La mediane est calculée avec
l'algorithme de complexité linéaire vu en cours: on
décompose le nuage de points en tableaux de taille 5, pour
lesquels on peut appeler la fonction précédente. Ensuite
on appelle récursivement la fonction findMedianLinear pour
calculer la médiane du nuage de points constitué des
medianes des points stockés dans les tableaux.
|

|

|
1.1b Calcul de la médiane de points déjà triés
|
voici quelque résultats
|
|
Pour le calcul de la médiane
on peut d'abord trier le nuage des points (par rapport à la
direction de coupe).
On vous demande de compléter la fonction suivante:
- private Point_d findMedian (PointCloud N, int cutDim)
qui renvoie la médiane d'un nuage de points (après avoir
trié les points selon la direction de coupe).
Suggestion: on
pourra se servir de la fonction auxiliaire
- private Point_d selectWithSorting (Point_D[] buffer,
int index, int cutDim)
qui renvoie le i-ième élément d'un tableau qu'il faut trier:
on pourra se servir de la méthode Arrays.sort() , qui
permet de trier un tableau d'objets, muni d'un comparateur
(voir classe CoordinateComparator).
|

 |
|
1.1c Utiliser le barycentre
|
Pour le calcul du plan
séparateur vous pouvez aussi vous servir du barycentre de
l'ensemble de points: dans ce cas vous n'avez rien à coder
pour l'instant... passez à l'exo suivant.
Rappel: le
barycentre d'un nuage de points est déjà codé par la méthode
PointCloud.mean(PointCloud
N).
|
1.2 Construction d'un Kd-Tree
Il vous reste maintenant à compléter le constructeur de la classe
KdTree, qui construit de manière
récursive un arbre correspondant à un nuage de points
donné.
Complétez le constructeur
- public KdTree (PointCloud N, int pDim, int cutDim)
Input:
- N: nuage de points
- pDim: dimension de l'espace où se trouvent les points
(aujourd'hui 2 ou 3)
- cutDim: direction de coupe (en entier entre 0 et d-1).
- Exemple: si cutDim=0, alors on est en train de couper avec
un hyper plan vertical parallèle aux axes, et orthogonale au
plan x=0.
Idée pour la solution: vous
pouvez vous inspirer des étapes suivantes:
Entrees: N, pDim, cutDim
Initialiser les données associées à un noeud donné du KdTree
- pDim, cutDim, points, numPoints
Calculer la valeur de coupe (cutValue)
- avec la mediane
- ou bien avec le barycentre
A partir du nuage N,
- creez deux sous listes lowerN et upperN: elles partitionnent la liste N
et correspondent aux points plus petits (ou plus grands) de la valeur cutValue
(selon la direction définie par cutDim)
Testez si jamais les deux sous-listes ne contiennent pas de points
- si jamais les sous-listes ne sont pas vides
alors créer des sous-arbres de manière récursive
(en passant les bons arguments, notamment la direction de coupe, qui devrait changer)
- si jamais les deux sous-listes sont vides (les deux)
retourner null (on est dans une feuille du Kd-Tree)
Pour tester, vous pouvez faire appel à la class TestKdTree.
Voici le résultat: (lecture du nuage de points dans 2D_dataset1.dat)
Exercice 1: testing Kd-trees
reading point cloud from file TD/input/dataset1.dat ... done
Drawing point cloud in 2D...
Warning: drawing in dimension 3 a cloud of points of dimension 2
visualizing 3D point cloud
exercice 1.2: constructing Kd-Tree
size original point cloud: 3000
size Kd-Tree (total number of nodes): 5999
number of leaves in the tree: 3000
Bad balance in kd-tree! (521/634=0.8217665615141956)
end
1.3 Range Search
Il vous reste maintenant à compléter la méthode suivante, qui
calcule manière récursive
les points à distance au plus sqRad d'un point donné.
- public PointCloud OrthognalRangeSearch (Point_D q, double
sqRad)
Input:
- q: point requete
- sqRad. rayon au carré, qui définit le disque où chercher les
voisins du point q.
Vous pouvez vous inspirer des étapes suivantes
OrthogonalRangeSearch(Nuage n, point q, distance R)
si on a atteint une feuille de l'arbre
retourner le point s'il est à distance R du point q (ou les points, si plusieurs)
sinon
tester si le disque intersecte ou pas le plan séparateur
- si le plan séparateur n'intersècte pas le disque (alors un seul des deux sous-noeud est à traverser)
trouver le sous-noeud S de l'arbre dont le rectangle correspondant touche le disque
appeler récursivement la fonction NN() sur le noeud S
- sinon (les deux sous-noeud sont à traverser)
appeler récursivement NN() sur les deux sous-arbres S1 et S2
soit L1=NN(S1)
soit L2=NN(S2)
retouner L1 union L2
Pour tester, vous pouvez faire appel à la classe TestRangeSearch
Voici le résultat avec java RangeSearch
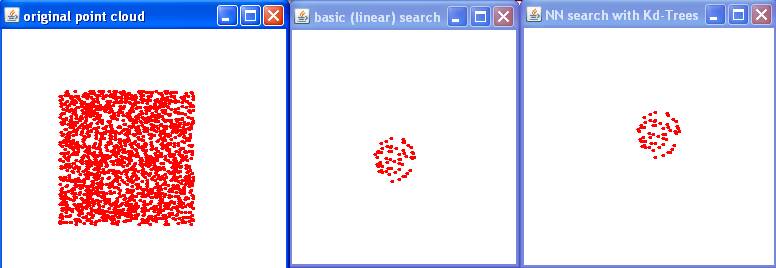
|
Exo 1.3: testing Range Search
Generated point cloud from random points in dimension 3
Drawing point cloud in 2D...
warning: wrong point dimension 3
visualizing 3D point cloud
Computing Kd-Tree data structure... done
visualizing 3D point cloud
basic search: 1165 neighbors
visualizing 3D point cloud
Kd-Trees search: 1165 neighbors
Range Search performaces:
Total construction time: 0s 32ms
Total NN query time: 0s 0ms
Testing Range Search: end
|
|
nuage
orginal
---
voisins calculés avec recherche
linéaire ---
voisins calculés avec Kd-Trees |
|
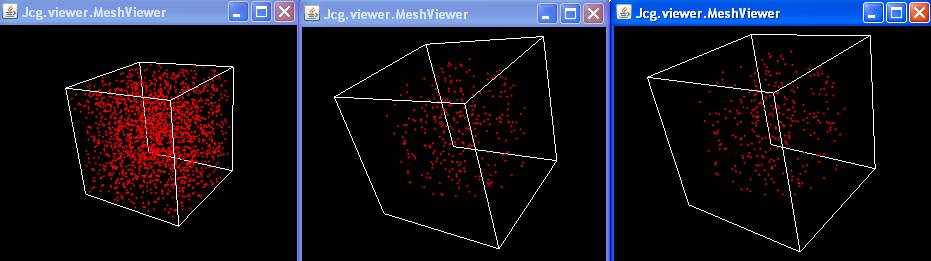
|
Exemple de range search en 3D
Nuage input: points aléatoires distribués uniformement dans
le carré unité
Output: voisins se trouvant dans une boule
Remarque:
les 3 images ne sont pas affichées à la meme échelle
|
Voici le résultat avec java RangeSearch dataset1.dat
Exo 1.3: testing Range Search
reading point cloud from file TD/input/dataset1.dat ... done
Drawing point cloud in 2D...
basic search: 43 neighbors
Warning: drawing in dimension 3 a cloud of points of dimension 2
visualizing 3D point cloud
Kd-Trees search: 43 neighbors
Range Search performaces:
Total construction time: 0s 31ms
Total NN query time: 0s 0ms
Testing Range Search: end
2. implementation de l'algorithme de clustering
Dans cet exercice on vous demande de compléter la classe MeanShiftClustering:
comme vu en cours aujourd'hui, l'algorithme Mean-Shift consiste à
classifier tous les points en entrés.
Remarque:
pour plus de détails vous pouvez consulter les transparents (page
web du cours)
Remarque (importante):
comment représenter la partition en clusters
La classe Cluster permet de décrire comment les
points sont associés aux clusters (une fois calculés). Pour se
faire, on utilise une table de hachage:
- HashMap<X,Integer>
clusters
qui permet d'associer un entier (Integer)
à chaque point en entrée (de type X):
il s'agit de l'indice i du cluster contenant le point,
lorsque le calcul a été effectué (i
prend des valeurs entre 0... C-1,
où C est le nombre de clusters)
La classe MeanShiftClustering contient
- N: nuage de points (input)
- clusters: table de hachage qui
décrit les clusters
- sqCvgRad: rayon de convergence (au
carré): égal à bandWidth*1e-3
- sqAvgRad: rayon de la Fenetre (au
carré): égal à bandWidth
- sqInflRad: rayon d'influence (au
carré): égal à bandWidth/4
- sqMergeRad: distance de fusion (au
carré): égal à bandWidth
- le constructeur public MeanShiftClustering
(PointCloud N, double bandWidth)
qui permet d'initialiser toutes les valeurs ci-dessus, en fonction
du paramètre bandwidth
(donné en input à l'algorithme).
Pour ce qui est du TD d'aujourd'hui, on suggère d'utiliser comme bandwidth
des valeurs réelles entre: 0.5 et 4.0
2.1 Detection d'un cluster
Il vous reste maintenant à compléter la méthode suivante, qui
calcule un cluster à partir d'un point donné seed.
- public PointCloud detectCluster (Point_D seed, int
clusterIndex)
Input:
- seed: centre de la fenetre initiale (en général, c'est un
point du nuage N, pas encore classifié)
- clusterIndex: on associe de numero au nouveau cluster
Output
- après le calcul, tous
les points associés au meme cluster doivent avoir été
classifiés: p.cluster=clusterIndex
- le résultat à la la sortie est une liste L (PointCloud)
contenant:
- les points appartenant au cluster
- le point stationnaire (au sommet de la liste): ce point
n'appartient pas en général au nuage N.
Pour tester, vous pouvez faire appel à la classe TestMeanShift
(fonction testDetectCluster(N,
bandWidth) dans le main)
Voici le résultat avec java MeanShiftAlgorithm
dataset1.dat 0.9
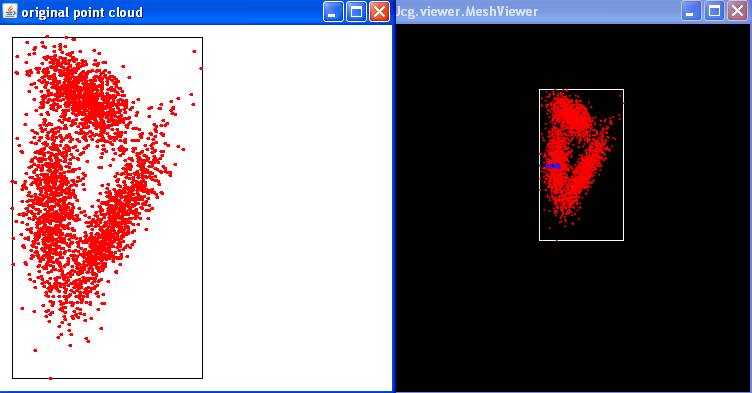
|
Exercice 2:
reading point cloud from file data/input/2D_dataset1.dat ...
done
point cloud of size: 3000
Drawing point cloud in 2D...
Exercice 2.1: testing detecting a cluster
size of the detected
cluster: 53
size
of the cluster: 52
Warning: drawing in dimension 3 a cloud of points of
dimension 2
visualizing 3D point cloud
Range Search performaces:
Total construction time: 0s 31ms
Total NN query time: 0s 0ms
Total time to find clusters: 0s 265ms
|
|
Sortie à la console
|
Voici le résultat avec java TestMeanShift 0.9
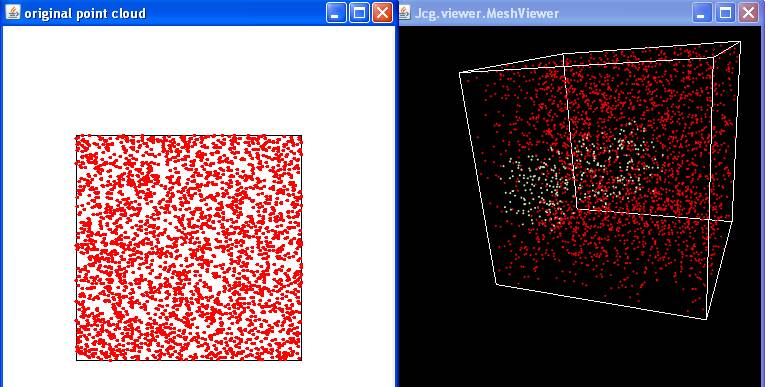
|
Exercice 2:
Generated point cloud from random
points
in dimension 3
point cloud of size: 3000
Drawing point cloud in 2D...
warning: wrong point dimension 3
Exercice 2.1: testing detecting a cluster
size
of the detected cluster: 315
size
of the cluster: 314
visualizing 3D point cloud
Range Search performaces:
Total construction time: 0s 31ms
Total NN query time: 0s 0ms
Total time to find clusters: 0s 281ms
|
|
Sortie à la console
|
2.2 Fusion de clusters
Dans cet exercice vous devez réaliser la fusion d'un cluster donné
avec un autre parmi ceux qui sont déjà existants: un cluster C1 est
fusionné avec un cluster Ci si les deux maximaux sont à distance au
plus R.
Complétez la fonction
- public int mergeCluster (PointCloud C1, Point_D[]
clusterCenters)
Input:
- C1: un nuage de points qu'il faut éventuellement
fusionner avec un cluster déjà existant
- Remarque: C1 contient c+1 points, car au sommet de la liste on
a stoqué le point stationnaire (le maximum local)
- clusterCenters: un tableau contenant les points stationnaires
(cluster centers) des clusters detectés jusqu'à une étape donnée
Output
- il faut fusionner
deux cluster si jamais le carré de leur distance est au plus sqMergeRad
(paramètre
de la classe MeanShidtClustering)
- le résultat est:
- -1: s'il n'y a pas de fusion
- i: si jamais C1 a été fusionné avec le cluster ayant point
stationnaire clusterCenter[i]
- à la sortie, il ne faut pas oublier de mettre à jour le champ cluster des
points dans C1.
Pas de test pour cette
fonction
3.3 Calcul de tous les clusters
Maintenant vous disposez de tous les outils pour détecter, de
manière itérative, tous les clusters.
Complétez la fonction
- public Point_D[] detectClusters ()
qui implémente l'algorithme Mean-Shift, en classifiant tous les
points du nuage original N (stoqué dans la classe
MeanShiftClustering )
Output
- il faut calculer
tous les clusters: tous les points de N doivent etre
partitionnés en clusters
- le résultat est un tableau de taille |N| contenant les
"cluster centers" de tous les clusters: si on a trouvé c
clusters alors
- les premières c cases du tableau sont non null
- les autres cases sont null
- à la sortie, le champ
cluster de tous les points de N doit etre un nombre non
négatif.
Suggestion:
vous pouvez vous inspirer des étapes suivantes
// initialiser la structure de donnees
- creer un tableau clusterCenters de type Point_D, de taille |N| (au debut les cases sont toutes null)
//(ce tableau contient les points stationnaires des clusters detectés, jusqu'à une certaine étape)
- initialiser une variable NbClusters=0, pour compter le nombre de clusters detectés
// procedure iterative de detection des clusters
Tant qu'il existe des points non classifiés
- choisir un point P (centre d'une Fenetre)
si jamais P est déjà classifié, on ne fait rien
sinon
- calculer le cluster associé à P // avec la fonction detectCluster()
soit la liste L(P) des points classifiés dans le cluster de P
- tester si L(P) peut etre fusionnée avec d'autres clusters déjà existants
(pour cela il on peut utilisé le vecteur clusterCenters)
- si on ne peut pas fusionner le cluster alors
ajouter ce cluster aux existants
// (ajouter le point stationnaire correspondant au tableu clusterCenters)
retourner comme sortie le tableau clusterCenters
Pour tester, vous pouvez faire appel à la classe TestMeanShift (fonction
testMeanShift(N,
bandWidth) dans le main)
Voici le résultat avec java MeanShiftAlgorithm
0.3
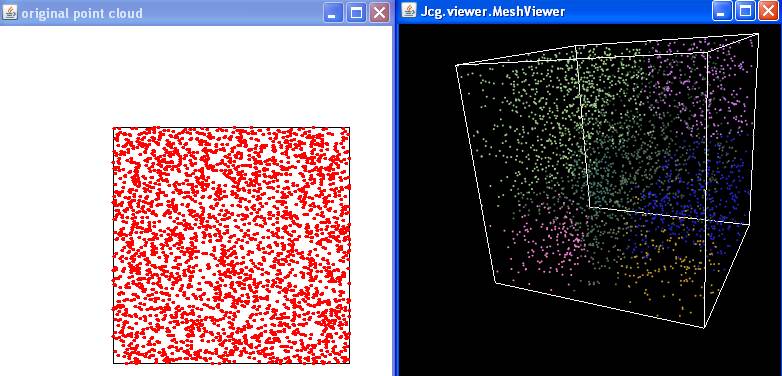 |
Exercice 2:
Generated point cloud from random points in dimension 3
point cloud of size: 3000
Drawing point cloud in 2D...
warning: wrong point dimension 3
Exercice 2.3: testing Mean-Shift clustering
Initializing data structure... done
Number of clusters detected: 9
visualizing 3D point cloud
Range Search performaces:
Total construction
time: 0s 32ms
Total NN query time: 2s 609ms
Total time to find
clusters: 3s 750ms
|
| nuage avant et après
clustering |
Sortie à la console
|
Voici le résultat avec java MeanShiftAlgorithm
dataset3.dat 1.2
 
|
Exercice 2.3: testing
Mean-Shift clustering
Initializing data structure... done
Number of clusters detected: 3
Warning: drawing in dimension 3 a cloud of points of
dimension 2
visualizing 3D point cloud
Range Search performaces:
Total construction
time: 0s 563ms
Total NN query time: 13s 791ms
Total time to find
clusters: 23s 47ms
|
dataset3.dat
|
Sortie à la console
|
3. clustering et segmentation d'images
Dans cet exercice on vous demande de tester avec la classe TestImageSegmentation.
Remarque:
vous n'avez plus rien à coder (ou presque): une fois l'algorithme de
clustering bien implementé, la segmentation d'image est effectuée
par la classe TestImageSegmentation, dont les méthodes
fournissent tout le nécéssaire pour obtenir, à partir d'un nuage de
points partitionné en clusters, l'image segmentée correspondante.
3.1 Tester l'algo de segmentation d'images (pas de code à
écrire)
Il ne vous reste qu'à faire des expéreinces sur les images de test,
à l'aide de la fonction main de la classe ImageSegmentation.
Afin de se rendre compte de l'utilité des Kd-Trees, vous pouvez
chercher à effectuer deux types de tests,
- en choisissant d'abord de localiser les plus proches voisins
de manière efficace (avec Kd-Trees),
- ou avec une simple recherche exhaustive (en temps
linéaire).
Pour cela il suffit de changer le choix de l'impl'ementation de
l'interface RangeSearch.
Voici nos images après segmentation, obtenues en executant par
exemple:
java TestImageSegmentation Steve.jpg 2.0
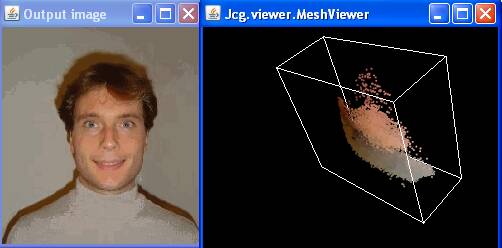
|

|
| Bandwidth 2.0, 51957
pixels, 608 detected clusters |
Bandwidth 2.0, 53280 pixels,
161 detected clusters |
efficient NN search (with
Kd-Trees)
Total time to find clusters: 21s 781ms
Range Search performaces:
Total construction time: 0s
953ms
Total NN query time: 15s
725ms
|
efficient NN search (with
Kd-Trees)
Total time to find clusters: 20s 437ms
Range Search performaces:
Total construction time: 0s
985ms
Total NN query time: 14s
405ms
|
basic
NN search (linear time search)
Total time to find clusters: 597s 375ms
Range Search performaces:
Total NN query time: 588s
893ms
|
basic NN search (linear time search)
Total time to find clusters: 311s 781ms
Range Search performaces:
Total NN query time:
304s 236ms
|
3.2 Apporter une petite amélioration
On va mettre en place une dernière petite optimisation, afin de réduire le temps de calcul
(et le nombre de cluster):
comme on peut le voir sur la sortie, le gain en termes de temps de
calcul est non negligeable, alors que le résultat final (la qualité
de la segmentation) est très proche de ce qu'on obtient sans
optimisation.
L'idée de base est très simple: à la fin de la procedure pour
detecter un clustering, après avoir atteint la convergence, on
ajoute au clustering tous les points qui se trouvent dans la fenetre
(le disque) dont le centre est le point stationnaire qu'on
vient de determiner.
Cela a pour effet d'augmenter "legerement" le nombre de points qui
appartiennent au cluster calculé: ce qui reduit le nombre de points
à classifier dans les étapes successives.
Modifiez la fonction
- public PointCloud detectCluster (Point_D seed, int
clusterIndex)
afin de mettre en place cette amélioration.
Voici quelque résultat

|

|
EP, 50700 pixels, bandwidth
2.5,
Detected clusters 1113...
without optimization
Total time to find clusters: 52s 406ms
Range Search performaces:
Total
construction
time: 1s 0ms
Total
NN query time: 35s 715ms
|
EP, 50700 pixels, bandwidth
2.5,
with optimization
Total time to find clusters: 43s 469ms
Range Search performaces:
Total
construction
time: 1s 31ms
Total
NN query time: 28s 758ms
|
































